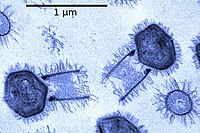
Vírus gigante
| Vírus gigante | |||||
|---|---|---|---|---|---|
| Classificação científica | |||||
| |||||
Um vírus gigante ou gigavírus é um vírus muito grande, alguns dos quais são maiores que as bactérias típicas[1][2][3]. Todos os vírus grandes e gigantes conhecidos até o momento pertencem ao filo Nucleocytoviricota[4]. Também são chamados de "vírus nucleocitoplasmáticos de grandes DNA" ou "nucleocytoplasmic large DNA viruses" (NCLDV), em inglês[5][6].
Esses vírus são excepcionais por possuírem genomas enormes em comparação com outros vírus, por possuírem muitos genes únicos não encontrados em outras formas de vida e, finalmente, por possuírem genes não associados com outros vírus, como genes auxiliadores do metabolismo de energia e de ácidos nucleicos[7][8][9][10].
Descrição
[editar | editar código-fonte]A exata definição de vírus gigantes é ainda discutida na literatura, mas, no geral, são considerados grandes ou gigantes aqueles vírus com capsídeos de formato pseudoicosaédrico entre 200 e 400 nanômetros, geralmente recobertos com uma camada de proteínas fibrosas. Esses vírus possuem genomas de DNA dupla fita (300 a >1000 quilo-pares de base) que codificam aproximadamente 1000 genes[11]. A maioria dos NCLDVs descritos até hoje estão nas famílias Mimiviridae.
Comparação dos maiores vírus gigantes conhecidos
[editar | editar código-fonte]| Nome gigante do vírus | Comprimento do genoma | Gene | Diâmetro do capsídeo (nm) | "Hair cover" | Genbank # |
|---|---|---|---|---|---|
| Bodo saltans virus[12] | 1,385,869 | 1227 proteínas (previstas) | ~300 | sim (~40 nm) | MF782455 |
| Megavirus chilensis[13] | 1,259,197 | 1120 proteínas (previstas) | 440 | sim (75 nm) | JN258408 |
| Mamavirus[14] | 1,191,693 | 1023 proteínas (previstas) | 500 | sim (120 nm) | JF801956 |
| Mimivirus[15][16] | 1,181,549 | 979 proteínas 39 não codificantes | 500 | sim (120 nm) | NC_014649 |
| Tupanvirus[17] | 1,500,000 | 1276-1425 proteínas | ≥450+550[18] | KY523104 MF405918[19] |
A lista inteira está na lista principal de vírus gigante criada pelo software Giant Virus Finder.[20]
| Nome gigante do vírus | Aminoacil-tRNA sintetase | Octocoral similar 1MutS | 2Portal estelar[21] | Virófago conhecido[22] | Fábrica de virion citoplasmático | Hospedeiro |
|---|---|---|---|---|---|---|
| Megavirus chilensis | 7 (Tyr, Arg, Met, Cys, Trp, Asn, Ile) | sim | sim | não | sim | Acanthamoeba (Unikonta, Amoebozoa) |
| Mamavirus | 4 (Tyr, Arg, Met, Cys) | sim | sim | sim | sim | Acanthamoeba (Unikonta, Amoebozoa) |
| Mimivirus | 4 (Tyr, Arg, Met, Cys) | sim | sim | sim | sim | Acanthamoeba (Unikonta, Amoebozoa) |
1Mutator S (MutS) e seus homólogos são uma família de proteínas de reparo de incompatibilidade de DNA envolvidas no sistema de reparo de incompatibilidade que atua para corrigir mutações pontuais ou pequenos loops de inserção/exclusão produzidos durante a replicação do DNA, aumentando a fidelidade da replicação. 2Um portal estelar é uma estrutura estelar de cinco pontas presente no capsídeo viral que forma o portal através do qual o núcleo interno da partícula é entregue ao citoplasma do hospedeiro[23].
O estudo de vírus gigantes no Brasil
[editar | editar código-fonte]O primeiro vírus gigante a ser propriamente descrito como um vírus gigante foi o Acanthamoeba polyphaga mimivirus (APMV), isolado na França, em 2003[24]. A partir disso, houve a curiosidade ao redor no mundo para encontrar outros vírus grandes e gigantes. No Brasil, não foi diferente[25], especialmente devido à ampla variedade de ambientes naturais encontradas no país, incluindo a Floresta Amazônica, o Cerrado, o Pantanal e a Caatinga.
Alguns dos vírus grandes e gigantes descritos no Brasil até hoje incluem:
- Tupanvirus altamarinense, prospectado a partir de sedimentos coletados a 3000 metros abaixo do nível do mar na Bacia de Campos, Rio de Janeiro[26];
- Tupanvirus salinum, isolado a partir de amostras coletadas de um lago hipersalino na Nhecolândia, Pantanal brasileiro[27];
- Sambavirus (SMBV), isolado a partir de amostras do Rio Negro, Manaus, Amazonas[28];
- Cedratvirus getuliensis, prospectado a partir de amostras de esgoto do estado de Minas Gerais[29];
- Fautovirus mariensis e Niemeyer virus, isolados da Lagoa da Pampulha, Belo Horizonte, Minas Gerais[30][31].
Todos esses vírus foram descritos pelo Grupo de Estudos de Vírus Gigantes, vinculado ao LabVírus, na Universidade Federal de Minas Gerais, Belo Horizonte, Minas Gerais, Brasil.
Referências
- ↑ Reynolds, Kelly A. (2010). «Mysterious Microbe in Water Challenges the Very Definition of a Virus» (PDF). Water Conditioning & Purification. Arquivado do original (PDF) em 19 de março de 2014
- ↑ Ogata, Hiroyuki; Kensuke Toyoda; Yuji Tomaru; Natsuko Nakayama; Yoko Shirai; Jean-Michel Claverie; Keizo Nagasaki (2009). «Remarkable sequence similarity between the dinoflagellate-infecting marine girus and the terrestrial pathogen African swine fever virus». Virology Journal. 6 (178). 178 páginas. PMC 2777158
 . PMID 19860921. doi:10.1186/1743-422X-6-178. Consultado em 30 de maio de 2011
. PMID 19860921. doi:10.1186/1743-422X-6-178. Consultado em 30 de maio de 2011
- ↑ Meet the giants among viruses The list of these mega-sized entities continues to grow por Emily Demarco (2018)
- ↑ «Giant Viruses». American Scientist (em inglês). 6 de fevereiro de 2017. doi:10.1511/2011.91.304. Consultado em 8 de agosto de 2024
- ↑ Colson P, de Lamballerie X, Fournous G, Raoult D (2012). «Reclassification of giant viruses composing a fourth domain of life in the new order Megavirales». Intervirology. 55 (5): 321–332. PMID 22508375. doi:10.1159/000336562
- ↑ Colson P, De Lamballerie X, Yutin N, Asgari S, Bigot Y, Bideshi DK, Cheng XW, Federici BA, Van Etten JL, Koonin EV, La Scola B, Raoult D (2013). «"Megavirales", a proposed new order for eukaryotic nucleocytoplasmic large DNA viruses». Arch Virol. 158 (12): 2517–21. PMC 4066373
 . PMID 23812617. doi:10.1007/s00705-013-1768-6
. PMID 23812617. doi:10.1007/s00705-013-1768-6
- ↑ Van Etten, James L. (Julho–Agosto de 2011). «Giant Viruses». American Scientist. 99 (4): 304–311. doi:10.1511/2011.91.304
- ↑ Zhang, Qi-Ya; Gui, Jian-Fang (13 de novembro de 2018). «Diversity, evolutionary contribution and ecological roles of aquatic viruses». Science China Life Sciences. 61 (12): 1486–1502. ISSN 1674-7305. doi:10.1007/s11427-018-9414-7
- ↑ Brahim Belhaouari, Djamal; Pires De Souza, Gabriel Augusto; Lamb, David C; Kelly, Steven L; Goldstone, Jared V; Stegeman, John J; Colson, Philippe; La Scola, Bernard; Aherfi, Sarah (8 de julho de 2022). Fouchier, Ron AM; van der Meer, Jos W, eds. «Metabolic arsenal of giant viruses: Host hijack or self-use?». eLife: e78674. ISSN 2050-084X. PMC PMC9270025
 Verifique
Verifique |pmc=(ajuda). PMID 35801640. doi:10.7554/eLife.78674. Consultado em 8 de agosto de 2024 - ↑ Moniruzzaman, Mohammad; Erazo Garcia, Maria Paula; Farzad, Roxanna; Ha, Anh D; Jivaji, Abdeali; Karki, Sangita; Sheyn, Uri; Stanton, Joshua; Minch, Benjamin (setembro de 2023). «Virologs, viral mimicry, and virocell metabolism: the expanding scale of cellular functions encoded in the complex genomes of giant viruses». FEMS Microbiology Reviews (5). ISSN 1574-6976. PMC PMC10583209
 Verifique
Verifique |pmc=(ajuda). PMID 37740576 Verifique|pmid=(ajuda). doi:10.1093/femsre/fuad053. Consultado em 8 de agosto de 2024 - ↑ «Giant Viruses». American Scientist (em inglês). 6 de fevereiro de 2017. Consultado em 8 de agosto de 2024
- ↑ Deeg CM, Chow CT, Suttle CA (março de 2018). «The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea». eLife. 7: e33014. PMC 5871332
 . PMID 29582753. doi:10.7554/eLife.33014
. PMID 29582753. doi:10.7554/eLife.33014
- ↑ Arslan D, Legendre M, Seltzer V, Abergel C, Claverie JM (outubro de 2011). «Distant Mimivirus relative with a larger genome highlights the fundamental features of Megaviridae». Proceedings of the National Academy of Sciences of the United States of America. 108 (42): 17486–91. Bibcode:2011PNAS..10817486A. PMC 3198346
 . PMID 21987820. doi:10.1073/pnas.1110889108
. PMID 21987820. doi:10.1073/pnas.1110889108
- ↑ Colson P, Yutin N, Shabalina SA, Robert C, Fournous G, La Scola B, Raoult D, Koonin EV (2011). «Viruses with more than 1,000 genes: Mamavirus, a new Acanthamoeba polyphaga mimivirus strain, and reannotation of Mimivirus genes». Genome Biology and Evolution. 3: 737–42. PMC 3163472
 . PMID 21705471. doi:10.1093/gbe/evr048
. PMID 21705471. doi:10.1093/gbe/evr048
- ↑ Raoult D, Audic S, Robert C, Abergel C, Renesto P, Ogata H, La Scola B, Suzan M, Claverie JM (novembro de 2004). «The 1.2-megabase genome sequence of Mimivirus». Science. 306 (5700): 1344–50. Bibcode:2004Sci...306.1344R. PMID 15486256. doi:10.1126/science.1101485
- ↑ Legendre M, Santini S, Rico A, Abergel C, Claverie JM (março de 2011). «Breaking the 1000-gene barrier for Mimivirus using ultra-deep genome and transcriptome sequencing». Virology Journal. 8 (1). 99 páginas. PMC 3058096
 . PMID 21375749. doi:10.1186/1743-422X-8-99
. PMID 21375749. doi:10.1186/1743-422X-8-99
- ↑ Abrahão J, Silva L, Silva LS, Khalil JY, Rodrigues R, Arantes T, Assis F, Boratto P, Andrade M, Kroon EG, Ribeiro B, Bergier I, Seligmann H, Ghigo E, Colson P, Levasseur A, Kroemer G, Raoult D, La Scola B (fevereiro de 2018). «Tailed giant Tupanvirus possesses the most complete translational apparatus of the known virosphere». Nature Communications. 9 (1). 749 páginas. Bibcode:2018NatCo...9..749A. PMC 5829246
 . PMID 29487281. doi:10.1038/s41467-018-03168-1
. PMID 29487281. doi:10.1038/s41467-018-03168-1
- ↑ head and tail, respectively
- ↑ soda lake and deep ocean species of Tupanvirues, respectively
- ↑ «Giant Virus Toplist». PIT Bioinformatics Group, Department of Computer Science. Eötvös University. 26 de março de 2015
- ↑ Zauberman N, Mutsafi Y, Halevy DB, Shimoni E, Klein E, Xiao C, Sun S, Minsky A (maio de 2008). Sugden B, ed. «Distinct DNA exit and packaging portals in the virus Acanthamoeba polyphaga mimivirus». PLoS Biology. 6 (5): e114. PMC 2430901
 . PMID 18479185. doi:10.1371/journal.pbio.0060114
. PMID 18479185. doi:10.1371/journal.pbio.0060114
- ↑ Fischer MG, Suttle CA (abril de 2011). «A virophage at the origin of large DNA transposons». Science. 332 (6026): 231–4. Bibcode:2011Sci...332..231F. PMID 21385722. doi:10.1126/science.1199412
- ↑ «Mystery of the giant virus' 'stargate' solved». Haaretz.com (em inglês). Consultado em 10 de junho de 2020
- ↑ Scola, Bernard La; Audic, Stéphane; Robert, Catherine; Jungang, Liang; de Lamballerie, Xavier; Drancourt, Michel; Birtles, Richard; Claverie, Jean-Michel; Raoult, Didier (28 de março de 2003). «A Giant Virus in Amoebae». Science (em inglês) (5615): 2033–2033. ISSN 0036-8075. doi:10.1126/science.1081867. Consultado em 8 de agosto de 2024
- ↑ Boratto, Paulo Victor M.; Serafim, Mateus Sá M.; Witt, Amanda Stéphanie A.; Crispim, Ana Paula C.; Azevedo, Bruna Luiza de; Souza, Gabriel Augusto P. de; Aquino, Isabella Luiza M. de; Machado, Talita B.; Queiroz, Victória F. (fevereiro de 2022). «A Brief History of Giant Viruses' Studies in Brazilian Biomes». Viruses (em inglês) (2). 191 páginas. ISSN 1999-4915. doi:10.3390/v14020191. Consultado em 8 de agosto de 2024
- ↑ Abrahão, Jônatas; Silva, Lorena; Silva, Ludmila Santos; Khalil, Jacques Yaacoub Bou; Rodrigues, Rodrigo; Arantes, Thalita; Assis, Felipe; Boratto, Paulo; Andrade, Miguel (27 de fevereiro de 2018). «Tailed giant Tupanvirus possesses the most complete translational apparatus of the known virosphere». Nature Communications. 749 páginas. ISSN 2041-1723. PMC 5829246
 . PMID 29487281. doi:10.1038/s41467-018-03168-1. Consultado em 8 de agosto de 2024
. PMID 29487281. doi:10.1038/s41467-018-03168-1. Consultado em 8 de agosto de 2024
- ↑ Abrahão, Jônatas; Silva, Lorena; Silva, Ludmila Santos; Khalil, Jacques Yaacoub Bou; Rodrigues, Rodrigo; Arantes, Thalita; Assis, Felipe; Boratto, Paulo; Andrade, Miguel (27 de fevereiro de 2018). «Tailed giant Tupanvirus possesses the most complete translational apparatus of the known virosphere». Nature Communications. 749 páginas. ISSN 2041-1723. PMC 5829246
 . PMID 29487281. doi:10.1038/s41467-018-03168-1. Consultado em 8 de agosto de 2024
. PMID 29487281. doi:10.1038/s41467-018-03168-1. Consultado em 8 de agosto de 2024
- ↑ Campos, Rafael K.; Boratto, Paulo V.; Assis, Felipe L.; Aguiar, Eric RGR; Silva, Lorena CF; Albarnaz, Jonas D.; Dornas, Fabio P.; Trindade, Giliane S.; Ferreira, Paulo P. (14 de maio de 2014). «Samba virus: a novel mimivirus from a giant rain forest, the Brazilian Amazon». Virology Journal (1). 95 páginas. ISSN 1743-422X. PMC PMC4113263
 Verifique
Verifique |pmc=(ajuda). PMID 24886672. doi:10.1186/1743-422X-11-95. Consultado em 8 de agosto de 2024 - ↑ Silva, Ludmila Karen dos Santos; Andrade, Ana Cláudia dos Santos Pereira; Dornas, Fábio Pio; Rodrigues, Rodrigo Araújo Lima; Arantes, Thalita; Kroon, Erna Geessien; Bonjardim, Cláudio Antônio; Abrahão, Jônatas Santos (5 de março de 2018). «Cedratvirus getuliensis replication cycle: an in-depth morphological analysis». Scientific Reports (em inglês) (1). 4000 páginas. ISSN 2045-2322. doi:10.1038/s41598-018-22398-3. Consultado em 8 de agosto de 2024
- ↑ Borges, Iara; Rodrigues, Rodrigo Araújo Lima; Dornas, Fábio Pio; Almeida, Gabriel; Aquino, Isabella; Bonjardim, Cláudio Antônio; Kroon, Erna Geessien; La Scola, Bernard; Abrahão, Jônatas Santos (15 de julho de 2019). Shisler, Joanna L., ed. «Trapping the Enemy: Vermamoeba vermiformis Circumvents Faustovirus Mariensis Dissemination by Enclosing Viral Progeny inside Cysts». Journal of Virology (em inglês) (14). ISSN 0022-538X. PMC PMC6600206
 Verifique
Verifique |pmc=(ajuda). PMID 31019058. doi:10.1128/JVI.00312-19. Consultado em 8 de agosto de 2024 - ↑ Boratto, Paulo V. M.; Arantes, Thalita S.; Silva, Lorena C. F.; Assis, Felipe L.; Kroon, Erna G.; La Scola, Bernard; Abrahão, Jônatas S. (10 de novembro de 2015). «Niemeyer Virus: A New Mimivirus Group A Isolate Harboring a Set of Duplicated Aminoacyl-tRNA Synthetase Genes». Frontiers in Microbiology (em inglês). ISSN 1664-302X. doi:10.3389/fmicb.2015.01256. Consultado em 8 de agosto de 2024